תערובת הגברה של TIANSeq HiFi
בְּמוֹרַד הַזֶרֶם
הגברת PCR ספריית NGS, הגברה PCR של הדור הראשון, רצף אבטחה, גילוי SNP, מוטציה ספציפית לאתר וכו '.
מאפיינים
■ הגברה ביעילות גבוהה: הקפידו על קצב ההמרה וצמצמו את מחזורי ההגברה.
■ עדיפות נמוכה: יעילות הגברה מאוזנת לתבניות DNA עם תוכן GC% שונה.
■ ספציפיות גבוהה: עם מאפיין HotStart וספציפיות חזקה.
■ נאמנות גבוהה: הנאמנות גבוהה פי 50 מהפולימראז של Taq DNA.
■ רגישות גבוהה: קלט התבנית יכול להיות עד 1 עמודים.
ניתן להתאים את כל המוצרים עבור ODM/OEM. לפרטים,אנא לחץ על שירות מותאם אישית (ODM/OEM)
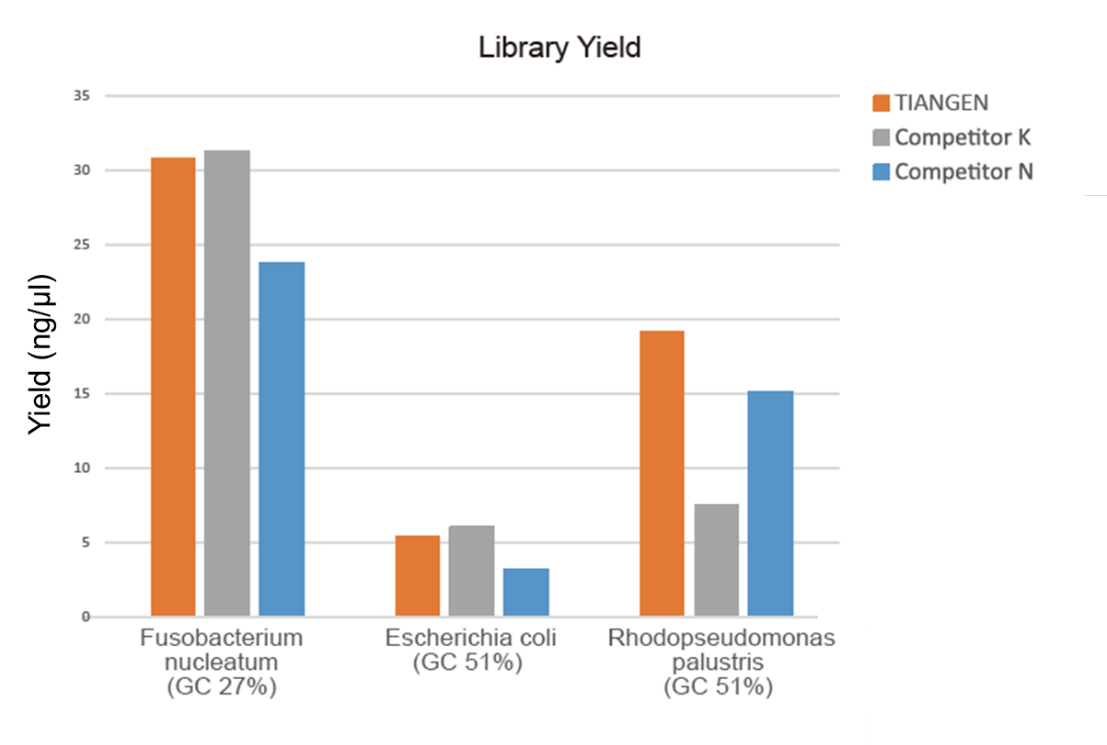 |
איור 1. העשרת הספרייה של DNA גנומי עם יחסי GC שונים (קלט גנומי 10 ng, 8 מחזורים הגברה) בוצעה בו זמנית באמצעות תערובת הגברה TIANSeq HiFi ואנזים HiFi מהספק K ו- N, ותשואת הספרייה זוהתה על ידי Agilent 2100. התוצאות הראו שלמיקס ההגברה של TIANSeq HiFi יש תפוקה גבוהה של ספרייה, עם אוניברסאליות חזקה לתכני GC שונים, וביצועי העשרת הספרייה היו טובים יותר מאשר ספקים אחרים. |
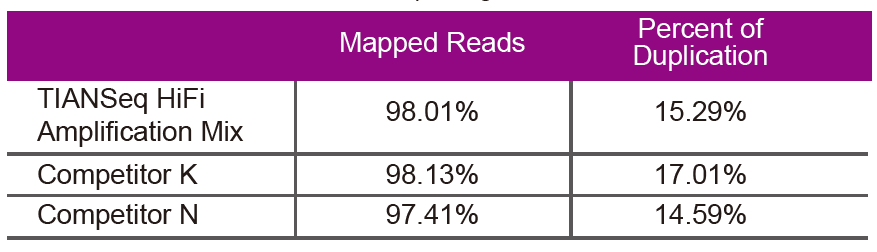 |
נתוני רצףהשתמש בתמהיל הגברת TIANSeq HiFi ואנזים HiFi המשמש במיוחד להגברת ספריית NGS מהספק K ו- N להגברת הספרייה של אותו ה- DNA הגנומי (קלט הגנום הוא 10 ng). לאחר רצף, נתח את הכיסוי ושיעור הכפילות של הספרייה. |
העדפת GC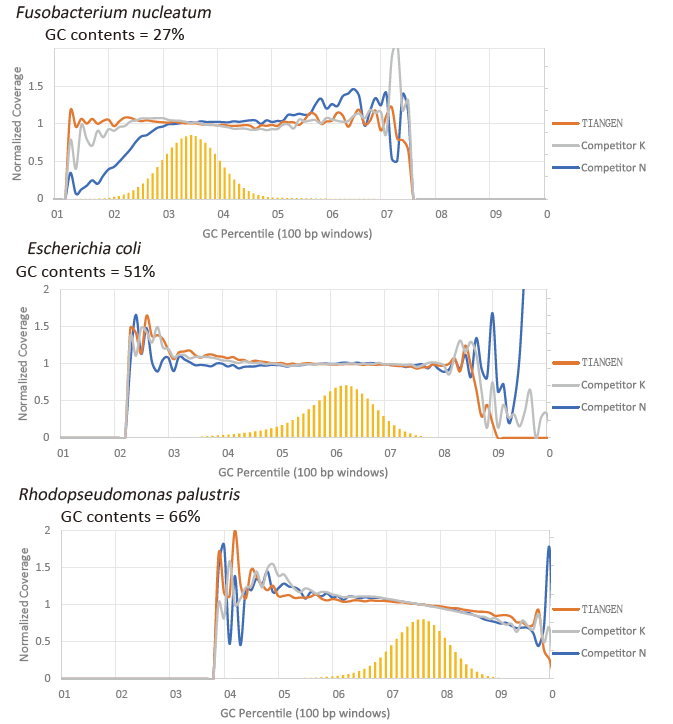 |
איור 2. הגבר ספריות הגנום עם תוכן CG שונה באמצעות תערובת הגברת TIANSeq HiFi ו- HIFi מהספק K ו- N. התוצאה מראה כי האחידות של ספריית הגברת תערובת TIANSeq HiFi Amplification היא טובה וללא העדפת GC, המקבילה לתוצאות של חברת K ומעט יותר מהמוצרים של חברת N. התוצאות מראות כי הכיסוי של ספריית הגברת תערובת ההגברה של TIANSeq HiFi הוא גבוה, קצב הכפילות עמד בדרישות וביצועי ההגברה של הספרייה שווים לזו של המתחרים. |
נכון לעכשיו, טכנולוגיית רצף תפוקה גבוהה מבוססת בעיקר על טכנולוגיית רצף הדור הבא. מכיוון שאורך הקריאה של טכנולוגיית הרצף של הדור הבא מוגבל, עלינו לפרק את רצף האורך המלא לספריות קטעים קטנים לרצף. על פי הצרכים של ניסויי רצף שונים, בדרך כלל אנו בוחרים רצף חד-קצוות או רצף כפול. כרגע שברי ה- DNA של ספריית רצפי הדור הבא מופצים בדרך כלל בטווח של 200-800 bp.

א) ה- DNA ירוד באיכותו ומכיל מעכבים. השתמש בדגימות DNA באיכות גבוהה כדי למנוע עיכוב של פעילות האנזים.
ב) כמות דגימת ה- DNA אינה מספיקה בעת שימוש בשיטה נטולת PCR לבניית ספריית DNA. כאשר הקלט של ה- DNA המקוטע עולה על 50 ng, ניתן לבצע סלקטיביות זרימת עבודה ללא PCR במהלך תהליך בניית הספרייה. אם מספר ההעתקה של הספרייה נמוך מכדי להיות ברצף ישיר, ניתן להגביר את ספריית ה- DNA על ידי PCR לאחר קשירת המתאם.
ג) זיהום RNA מוביל לכימות ה- DNA הראשונית הלא מדויקת זיהום ה- RNA עשוי להתקיים בתהליך הטיהור של ה- DNA הגנומי, מה שעלול להוביל לכימות DNA לא מדויקת ולהעמסת DNA לא מספקת במהלך בניית הספרייה. ניתן להסיר RNA על ידי טיפול ב- RNase.
א -1
א) מופיעים שברים קטנים (60 bp-120 bp) שברים קטנים הם בדרך כלל שברי מתאם או דימרים שנוצרים על ידי מתאמים. טיהור עם חרוזים מגנטיים של Agencourt AMPure XP יכול להסיר ביעילות שברי מתאם אלה ולהבטיח איכות רצף.
ב) שברים גדולים מופיעים בספרייה לאחר הגברת PCR. גודל שבר ה- DNA של הספרייה יגדל ב- 120 bp לאחר חיבור המתאם. אם שבר ה- DNA גדל ביותר מ -120 bp לאחר קשירת המתאם, הוא עשוי להיגרם על ידי הגברה לא תקינה של שבר של הגברת PCR מוגזמת. צמצום מספר מחזורי ה- PCR יכול למנוע את המצב.
ג) גודל חריג של שברי DNA הספרייה לאחר קשירת מתאם אורך המתאם בערכה זו הוא 60 bp. כאשר שני קצוות השבר נקשרים למתאמים, האורך רק יגדל ב- 120 bp. בעת שימוש במתאם אחר מזה שסופק על ידי ערכה זו, אנא צור קשר עם הספק למסור מידע רלוונטי כגון אורך המתאם. ודא שזרימת העבודה והתפעול של הניסוי בצע את השלבים המתוארים במדריך.
ד) גודל שברי DNA לא תקין לפני קשירת המתאם הסיבה לבעיה זו עלולה להיגרם כתוצאה מתנאי תגובה שגויים במהלך פיצול ה- DNA. יש להשתמש בזמני תגובה שונים עבור קלט DNA שונה. אם קלט ה- DNA הוא יותר מ -10 ng, אנו ממליצים לבחור את זמן התגובה של 12 דקות כזמן ההתחלה לאופטימיזציה, וגודל השבר המיוצר בשלב זה הוא בעיקר בטווח של 300-500 bp. משתמשים יכולים להגדיל או להקטין את אורך שברי ה- DNA למשך 2-4 דקות בהתאם לדרישות שלהם כדי לייעל את שברי ה- DNA בגודל הנדרש.
א -2
א) זמן הפיצול אינו מותאם אם ה- DNA המקוטע קטן מדי או גדול מדי, עיין בהנחיות לבחירת זמן פיצול המפורט בהוראה לקביעת זמן התגובה, והשתמש בנקודת זמן זו כבקרה, בנוסף הגדר מערכת התגובה להאריך או לקצר 3 דקות כדי לבצע התאמה מדויקת יותר בזמן הפיצול.
A-3
התפלגות גודל חריגה של ה- DNA לאחר טיפול בפיצול
א) שיטת הפשרה לא נכונה של ריאגנט פיצול, או שהריאגנט אינו מעורבב לחלוטין לאחר ההפשרה. להפשיר את מגיב תערובת האנזים 5 × פיצול על קרח. לאחר ההפשרה, מערבבים את המגיב באופן שווה על ידי החלקה עדינה בתחתית הצינור. אין לסובב את המגיב!
ב) דגימת קלט ה- DNA מכילה EDTA או מזהמים אחרים הידלדלות יוני מלח וסוכני chelating בשלב טיהור ה- DNA חשובה במיוחד להצלחת הניסוי. אם ה- DNA מומס ב 1 × TE, השתמש בשיטה המופיעה בהוראה לביצוע פיצול. אם ריכוז ה- EDTA בתמיסה אינו ודאי, מומלץ לטהר את ה- DNA ולמוססו במים לא יונים לתגובה שלאחר מכן.
ג) כימות DNA ראשונית לא מדויקת גודל ה- DNA המקוטע קשור קשר הדוק לכמות קלט ה- DNA. לפני טיפול בפיצול, כימות מדויקת של DNA באמצעות Qubit, Picogreen ושיטות אחרות חיונית לקביעת הכמות המדויקת של ה- DNA במערכת התגובה.
ד) הכנת מערכת התגובה אינה פועלת בהתאם להוראות הכנת מערכת התגובה המקוטעת חייבת להתבצע על קרח אך ורק בהתאם להוראות. כדי להבטיח את האפקט הטוב ביותר, יש להניח את כל רכיבי התגובה על קרח ולהכין את מערכת התגובה לאחר קירור מלא. לאחר השלמת ההכנה, אנא העלה או פיפטה לערבב היטב. אין לסובב!
1. שיטת ערבוב לא תקינה (מערבולת, תנודה אלימה וכו ') תגרום להפצה לא תקינה של שברי ספריות (כפי שמוצג באיור הבא), ובכך תשפיע על איכות הספרייה. לכן, בעת הכנת פתרון תגובת תערובת השבר, אנא העביר בעדינות פיפטה למעלה ולמטה כדי לערבב, או השתמש בקצה האצבע כדי לנענע ולערבב באופן שווה. היזהר לא לערבב עם מערבולת.

2. יש להשתמש ב- DNA בטוהר גבוה לבניית ספריה
■ תקינות DNA טובה: רצועת האלקטרופורזה היא יותר מ -30 קילוגרם, ללא זנב
■ OD260/230:> 1.5
■ OD260/280: 1.7-1.9
3. כמות הקלט של ה- DNA חייבת להיות מדויקת. מומלץ להשתמש בשיטות Qubit ו- PicoGreen כדי לכמת DNA, ולא ב- Nanodrop.
4. יש לקבוע את תכולת EDTA בתמיסת ה- DNA ל- EDTA יש השפעה רבה על תגובת הפיצול. אם התוכן של EDTA גבוה, יש לבצע טיהור DNA לפני הבדיקה הבאה.
5. יש להכין את פתרון תגובת הפיצול על קרח. תהליך הפיצול רגיש לטמפרטורת התגובה ולזמן (במיוחד לאחר הוספת משפר). על מנת להבטיח את דיוק זמן התגובה, אנא הכינו את מערכת התגובה על קרח.
6. זמן תגובת הפיצול חייב להיות מדויק זמן התגובה של שלב הפיצול ישפיע ישירות על גודל מוצרי השברים, ובכך ישפיע על התפלגות הגודל של שברי DNA בספרייה.
1. איזה סוג מדגם ישים לערכה זו?
סוג המדגם הרלוונטי של ערכה זו יכול להיות RNA כולל או mRNA מטוהר עם תקינות RNA טובה. אם משתמשים ב- RNA הכולל לבניית הספרייה, מומלץ להשתמש בערכת דלדול rRNA (Cat#4992363/4992364/4992391) כדי להסיר rRNA תחילה.
2. האם ניתן להשתמש בדגימות FFPE לבניית ספרייה עם ערכה זו?
ה- mRNA בדגימות FFPE יופחת במידה מסוימת, עם שלמות ירודה יחסית. בעת שימוש בערכה זו לבניית הספרייה, מומלץ לייעל את זמן הפיצול (לקצר את זמן הפיצול או לא לבצע פיצול).
3. באמצעות שלב בחירת הגודל המופיע במדריך המוצר, מה עלול לגרום לקטע המוכנס להראות סטייה קלה?
בחירת המידות תתבצע בהתאם להקפדה על שלב בחירת המידות במדריך המוצר הזה. אם יש סטייה, הסיבה עשויה להיות שהחרוזים המגנטיים אינם מאוזנים לטמפרטורת החדר או אינם מעורבים במלואם, הפיפטה אינה מדויקת או שהנוזל נשאר בקצה. מומלץ להשתמש בטיפים עם ספיגה נמוכה לניסוי.
4. בחירת מתאמים בבניית ספריה
ערכת הבנייה של הספרייה אינה מכילה ריאגנט למתאם, ומומלץ להשתמש בערכה זו יחד עם מתאם אינדקס יחיד של TIANSeq (Illumina) (4992641/4992642/4992378).
5. QC של הספרייה
זיהוי כמותי של הספרייה: Qubit ו- qPCR משמשים לקביעת ריכוז המסה והריכוז הטוחני של הספרייה בהתאמה. הפעולה היא אך ורק בהתאם למדריך המוצר. ריכוז הספרייה יענה בדרך כלל על הדרישות של רצף NGS. זיהוי טווח הפצת הספרייה: שימוש ב- Agilent 2100 Bioanalyzer לאיתור טווח הפצת הספרייה.
6. בחירת מספר מחזור ההגברה
על פי ההוראות, מספר מחזורי ה- PCR הוא 6-12, ויש לבחור את מספר מחזורי ה- PCR הדרושים בהתאם לקלט המדגם. בספריות בעלות תשואה גבוהה, הגברה יתר מתרחשת בדרך כלל בדרגות שונות, המתבטאות בשיא מעט גדול יותר לאחר שיא טווח היעד באיתור Agilent 2100 Bioanalyzer, או שהריכוז שזוהה של Qubit נמוך מזה של qPCR. הגברה קלה היא תופעה נורמלית, שאינה משפיעה על רצף הספרייה וניתוח הנתונים לאחר מכן.
7. קוצים מופיעים בפרופיל האיתור של Agilent 2100 Bioanalyzer
הופעת קוצים בזיהוי ביו -אנאליזר Agilent 2100 נובעת מהפיצול הלא אחיד של הדגימות, שם יהיו יותר שברים בגודל מסוים, וזה יתברר יותר לאחר העשרת PCR. במקרה זה, מוצע שלא לבצע את בחירת הגודל, כלומר להגדיר את מצב הפיצול ל- 94 ° C למשך 15 דקות מודגרות, כאשר חלוקת השברים קטנה ומרוכזת, וניתן לשפר את ההומוגניות.
קטגוריות מוצרים
מדוע לבחור בנו
מאז הקמתו, המפעל שלנו מפתח מוצרים ברמה עולמית ראשונה תוך הקפדה על העיקרון
של איכות קודם כל. המוצרים שלנו צברו מוניטין מעולה בתעשייה ואמינות ערך בקרב לקוחות חדשים וישנים.








